همکاری در وبلاگ
در جهت بالابردن و تقویت منابع آموزشی و علمی وبلاگ از دوستانی که تمایل به همکاری در این وبلاگ دارند دعوت میشود.
جهت همکاری با ایمیل زیر یا تلگرام مکاتبه فرمایید:
با تشکر
مدیریت وبلاگ
در جهت بالابردن و تقویت منابع آموزشی و علمی وبلاگ از دوستانی که تمایل به همکاری در این وبلاگ دارند دعوت میشود.
جهت همکاری با ایمیل زیر یا تلگرام مکاتبه فرمایید:
با تشکر
مدیریت وبلاگ
انجام انواع آنالیزهای آماری با نرم افزارهای مختلف R و SAS, Minitab, SPSS و ....
تجزیه واریانس (ANOVA) یک متغیره، چند متغیره (MANOVA)، مقایسات میانگین، رگرسیون، همبستگی و ..
تجزیه داده های مولکولی، ژنوتیپی و ...
ویرایش گرامری، اصلاح (Proofreading)، پارافریز کردن مقالات علمی جهت ارسال به ژورنال
09183416457

PAST نرم افزار کم حجم و در عین حال جامعی برای انجام انواع مختلف آنالیزهای آماری یک متغیره و چندمتغیره و ترسیم نمودارهای گوناگون می باشد. از کاربردهای اختصاصی این نرم افزار، آنالیز داده های اکولوژیکی، برآورد شاخص های تنوع، سری های زمانی و تحلیل مکانی ، مورفومتری و چینه نگاری است.
این نرم افزار رایگان را می توانید از سایت اصلی آن دانلود کنید:

Primer Premier یکی از جامع ترین ابزارهای طراحی بهینه پرایمر برای PCR، تعیین توالی و هیبریداسیون است. پرایمرها میتوانند به صورت خودکار یا دستی طراحی شوند. از ویژگی های این نرم افزار می توان به موارد زیر اشاره کرد:
- طراحی پرایمر برای تکثیر توالی ها از چندین گونه و پرایمرهای شناسایی پاتوژن
- طراجی پرایمرهای اختصاصی آلل
- طراحی پرایمرهای Degenerate
- طراحی پرایمرهای Nested/multiplex
- استفاده از آنزیم های برشی و آنالیز موتیف
- همردیف کردن توالی ها با استفاده از ClustalW
- همردیف کردن چندگانه توالی ها
- Long PCR
و بسیاری قابلیت های دیگر
برای دریافت نسخه کامل این نرم افزار با ایمیل یا شماره زیر مکاتبه فرمایید (قیمت 50 هزار تومان)
09183416457

Clone Manager Professional نرم افزار جامعی شامل مجموعه ای از ابزارها برای کلون سازی، برش ها و اتصالات آنزیمی، همردیف کردن توالی ها، یافتن تشابهات، طراحی پرایمر و .... می باشد.
از جمله قابلیت های این نرم افزار می توان به موارد زیر اشاره کرد:
- نمایش و ویرایش فایل های توالی
- یافتن نقاط برش آنزیم ها و نمایش اطلاعات آنزیم و انجام آنالیز برش آنزیمی
- برش، اتصال DNA و تغییر انتهای مولکول
- کلون سازی Gateway و TOPO
- ترسیم نقشه های گرافیکی با کیفیت
- جستجو برای پرایمرها، آنالیز آن ها و BLAST پرایمرها
- یافتن ORF
- ترجمه ژن ها
و بسیاری قابلیت های دیگر
برای دریافت نسخه کامل این نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)
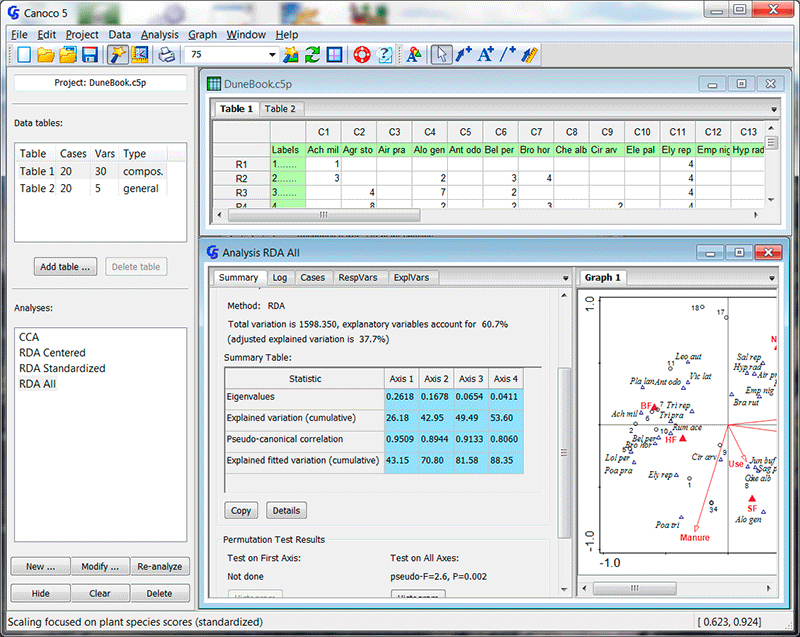
کانوکو (Canoco) یکی از محبوبترین نرم افزارها برای انالیزهای آماری چند متغیره با استفاده از روش های دسته بندی (Ordination) در رشته های اکولوژی است. این برنامه روش هایی متنوع و جدیدی در دسته بندی، از جمله تقسیم تنوع (variation partitioning)، تحلیل هم تناظری (co-correspondence analysis)، تحلیل افزونگی مبتنی بر فاصله (distance-based redundancy analysis) و دسته بندی محدود دوگانه (double constrained ordination) برای ارتباط صفت-محیط را می تواند انجام دهد.
از قابلیت های مهم این نرم افزار می توان به موارد زیر اشاره کرد:
- وارد کردن فایل داده اکسل بصورت مستقیم و ویرایش آن
- انجام روش های DCA، CA، CCA، DCCA، PCA و RDA
- آزمون های جایگشت مونته کارلو
- ابزارهای نمایش متعدد (مدل های Loess ، GLM و GAM برای نمایش خصوصیات داده روی فضای دسته بندی)
- معیارهای فاصله متعدد ( ضرایب Bray-Curtis، Gower، Jaccard و ...)
- گزینش گام به گام متغیرهای پیشگویی کننده
و ...
برای دریافت این نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)

معرفی دو نرم افزار کاربردی در تحقیقات کشاورزی ارائه شده توسط سیمیت (CIMMYT)
- META-R (Multi Environment Trail Analysis with R for Windows):
این نرم افزار شامل مجموعه ای از برنامه های R برای تجزیه های آماری است که از جمله کارکردهای آن می توان به موارد زیر اشاره کرد:
محاسبه پارامترهای BLUE و BLUP، همبستگی های ژنتیکی بین مکان ها و همبستگی های ژنتیکی بین متغیرها، وراثت پذیری عمومی و دیگر آماره ها برای آزمایشات اصلاحی، ساخت نمودارهای جعبه ای و هیستوگرام. امکان انجام آنالیزها بر اساس مکان، در بین شرایط محیطی و در بین همه مکان ها وجود دارد. این نرم افزار دارای رابط گرافیکی جاوا می باشد که به کاربر کمک می کند به راحتی بتواند فایل های ورودی داده ها، نوع آنالیز مد نظر و متغیرهای مورد بررسی را انتخاب کند.
دانلود META-R:
https://data.cimmyt.org/api/access/datafile/9090?gbrecs=true
- BIO-R (Biodiversity analysis with R for Windows):
این نرم افزار شامل مجموعه ای از برنامه های R است که آنالیز تنوع زیستی داده های مولکولی را انجام می دهد. از جمله کارکردهای آن می توان به موارد زیر اشاره کرد:
محاسبه هتروزیگوسیتی، تنوع بین و درون گروه ها، شاخص شانون، تعداد الل موثر، درصد لوکوس های پلی مورفیک، فاصله های ژنتیکی Rogers و Nei، تجزیه کلاستر، مقیاس بندی چند بعدی با پلات های دو بعدی و سه بعدی و ...
دانلود BIO-R:
https://data.cimmyt.org/api/access/datafile/9087?gbrecs=true
- ADEL-R (Analysis and design of experiments with R for Windows):
این نرم افزار شامل مجموعه ای از برنامه های R است که برای ایجاد و آنالیز طرح های آزمایشی کاربرد دارد. خروجی آن جدول های تجزیه واریانس (ANOVA) و گراف های مقایسه میانگین است. طرح هایی مانند طرح بلوک های کامل تصادفی، طرح بلوک های ناقص طرح آگمنت ( حجیم شده)، طرح کرت های خرد شده، طرح های فاکتوریل، طرح کرت های نواری. این نرم افزار دارای رابط گرافیکی جاوا است که استفاده آسان از آن را برای کاربر میسر می سازد.
دانلود ADEL-R:
https://data.cimmyt.org/api/access/datafile/9569?gbrecs=true

Plymouth Routines In Multivariate Ecological Research) PRIMER) یک پکیج آماری است که شامل مجموعه ای از روش های تخصصی یک متغیره، چند متغیره برای آنالیز داده های حاصل از نمونه گیری گونه ها است که در تحقیقات اکولوژی کاربرد فروان دارند. این روش ها شامل موارد زیر می شوند:
- گروه بندی سلسله مراتبی نمونه ها (CLUSTER)
- مقیاس بندی چند بعدی (MDS) غیر متریک
- تجزیه به مولفه های اصلی (PCA)
- آزمون تشابهات (ANOSIM) مبتنی بر جایگشت
- آنالیز درصد تشابه (SIMPER) به منظور شناسایی گونه های نشان دهنده تفرق بین دو گروه
- همبستگی و ارتباط الگوهای زیستی چند متغیره با مجموعه ای از متغیرهای محیطی (BEST و LINKTREE)
- آزمون های مقایسه ای مانند مانتل بر روی ماتریس های تشابه (RELATE)
- اندیس های تنوع استاندارد ، پلات های غالبیت، توزیع های هندسی فراوانی
و ...
نسخه آزمایشی این نرم افزار را می توانید از لینک زیر دانلود نمایید.
https://www.dropbox.com/s/c8xptra06kxatin/PRIMER6_1_8.msi?dl=0
برای دریافت لایسنس این نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)

امروزه یکی از نرم افزارهایی که کاربرد بسیار زیادی برای آنالیز داده ها دارد، نرم افزار R است. R یک زبان برنامه نویسی و همچنین نرم افزاری برای انجام آنالیزهای آماری ساده و پیچیده است که شامل عملکردهایی برای خلاصه سازی داده ها، آنالیز، نمایش گرافیکی و مدل سازی داده ها است. زبان R اولین بار توسط دو نفر در دانشگاه Auckland نیوزیلند پا به عرصه وجود گذاشت. اولین نسخه آن در سال 2000 ارایه شد. یک تیم بین المللی مسئولیت نگه داری، پشتیبانی و توسعه آن را به عهده دارد.
این نرم افزار را می توان بصورت رایگان از سایت اختصاصی آن دانلود کرد:
قصد داریم به مرور آموزش های عملی برای استفاده از این نرم افزار در تجزیه داده های ژنتیکی در وبلاگ قرار دهیم که می تواند مورد استفاده محققین و دانشجویان گرامی باشد.

این کتاب یکی از بهترین و جامع ترین کتاب ها برای تجزیه های آماری و ژنتیکی با استفاده از نرم افزارهای مختلف بخصوص برای دانشجویان و محققین رشته های کشاورزی، منابع طبیعی، زیست شناسی است. از مزایای این کتاب می توان به انجام مرحله به مرحله تجزیه ها بصورت تصویری اشاره کرد.
نرم افزارهایی که در این کتاب تدریس شده اند(همانطور که روی جلد کتاب هم ذکر شده است) شامل موارد زیر است:
SAS
SPSS
GenStat
PAST
GeneALEx
PopGen
MEGA
Structure
JoinMap
GGE-Biplot
Minitab
MSTAT-C
Path74
NTSys
DARwin
Oligo
Tassel
QTLcart
MapChart
Power Marker
کتاب را می توانید از سایت انتشارات دانشگاه تهران سفارش و تهیه نمایید:
فایلهای ضمیمه این کتاب نیز از طریق لینک زیر قابل دریافت است:
http://press.ut.ac.ir/data/ut/news/325.rar

جزوه زیر نسخه اول از راهنمای عملی تجزیه و تحلیل داده های مولکولی با استفاده از نرم افزارهای NTSys، DARwin و MEGA است که گردآوری و تدوین کردم.
امیدوارم مورد استفاده علاقه مندان قرار بگیرد.

کتاب بسیار خوب و جامعی درباره آنالیزهای بیوانفورماتیک است که به زبان ساده و با تصاویر، آنالیزهای متعددی بر روی DNA، RNA , پروتئین رو آموزش داده است (سال انتشار 2017).
این کتاب رو می توانید بطور رایگان از لینک زیر دریافت نمایید:
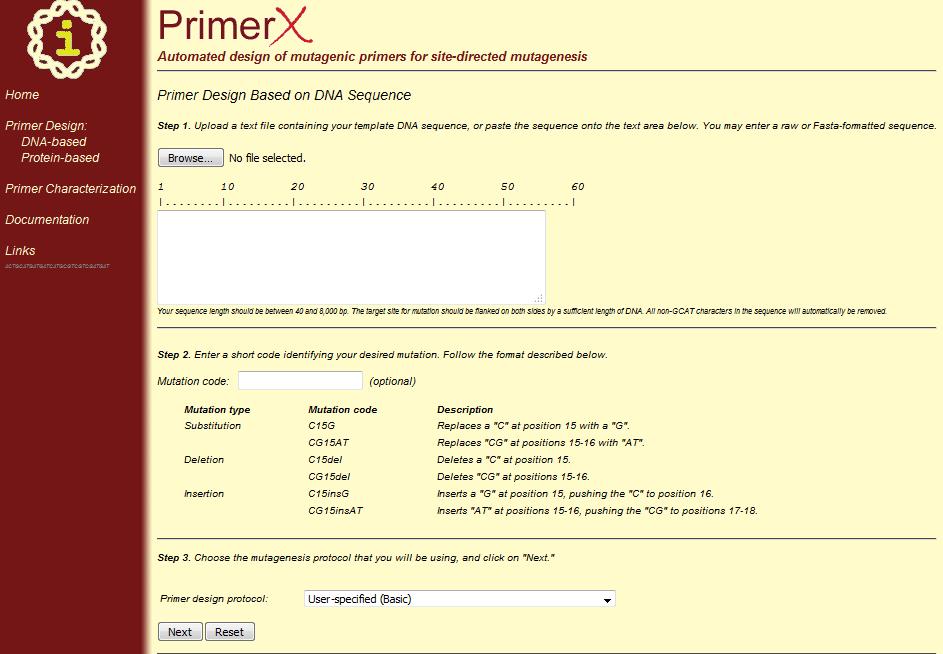
PrimerX یک برنامه مبتنی بر وب (آنلاین) است که امکان طراحی اتوماتیک پرایمرهای PCR موتاژنیک (جهش زا) را برای تکنیک جهش زایی هدفمند (site-directed mutagenesis) فراهم می کند. بر اساس اطلاعات ورودی شما، این برنامه یک توالی DNA الگو را با یک توالی DNA یا پروتین که از قبل جهش مورد نظر را تلفیق می سازد، مقایسه می کند. این برنامه طراحی جفت پرایمرهای موتاژنیک را بر اساس دو نوع توالی مختلف انجام می دهد. یکی وارد نمودن یک جهش در توالی DNA الگوی شما است بطوریکه درج ها (insertions)، حذف ها (deletions) و یا جایگزین های (substitutions) جفت باز مطلوب تلفیق می شوند. این مورد برای تولید SNP های اختصاصی و Indel ها توصیه می شود. دومی وارد نمودن یک جهش درون توالی پروتئین کد شده بوسیله DNA الگوی شما است که در این حالت این برنامه پرایمرهای موتاژنیک را بر اساس همه توالی های DNA ممکن که می توانند جهش مورد نظر را کد کنند، می سازد. این مورد برای تغییر دادن یک آمینواسید خاص به دیگری توصیه می شود. همچنین این برنامه می تواند پرایمرهایی که طراحی شده اند را بررسی و خصیصه یابی نماید. در این حالت تنها شما بایستی توالی پرایمر موتاژنیک و تعداد بازهای جفت نشدنی (mismatched bases) را وارد نمایید و خود برنامه محاسبات و گزارش رشته مکمل معکوس، محتوای GC، دمای ذوب و غیره را به شما ارائه می کند.
برای استفاده از این برنامه آنلاین به لینک زیر مراجعه نمایید:
https://www.bioinformatics.org/primerx/index.htm
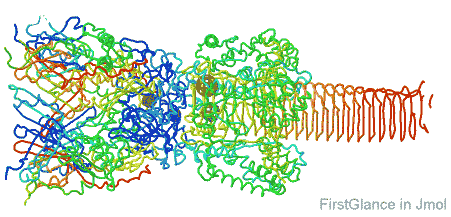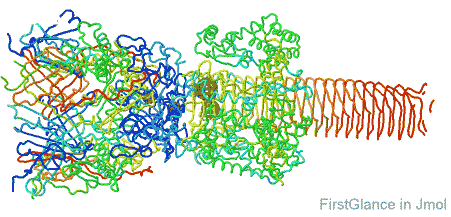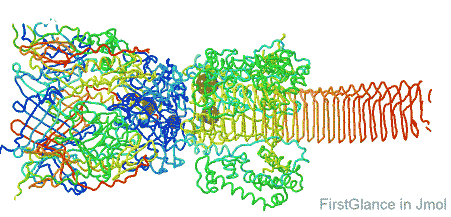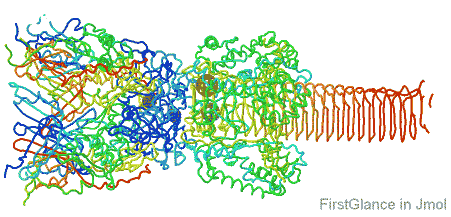

FirstGlance in Jmol ابزار ساده و سریعی برای مشاهده ساختار سه بعدی (3D) ماکرومولکول ها (پروتئین ها، DNA، RNA) و ترکیباتشان است. این ابزار مبتنی بر وب بوده و نیاز به نصب نرم افزار Jmol و یادگیری دستورات آن ندارد. برای استفاده از این ابزار، بایستی وارد لینک زیر شوید و کد شناسایی چهار رقمی مولکول مورد نظر که در پایگاه اطلاعاتی PDB موجود است را وارد نمایید.
http://bioinformatics.org/firstglance/fgij/
این نرم افزار با دارا بودن کنترل پنل های مختلف قابلیت های بسیار متنوعی از جمله نمایش اطلاعات جامعی از مدل مولکول (رزولوشن، زنجیره ها و توالی، تعداد و نوع عناصر و اتم ها و ...)، ابزارهایی برای نمایش پیوندهای دی سولفیدی و غیر کووالانسی، اندازه گیری فواصل و زاویه ها و .... فراهم می کند.
ویژگی مثبت این نرم افزار این است که بطور مداوم بروزرسانی می شود و همچنین در حالت پیش فرض بصورت غیر جاوا است و بنابراین می تواند روی گوشی های هوشمند و iPad ها هم اجرا شود.
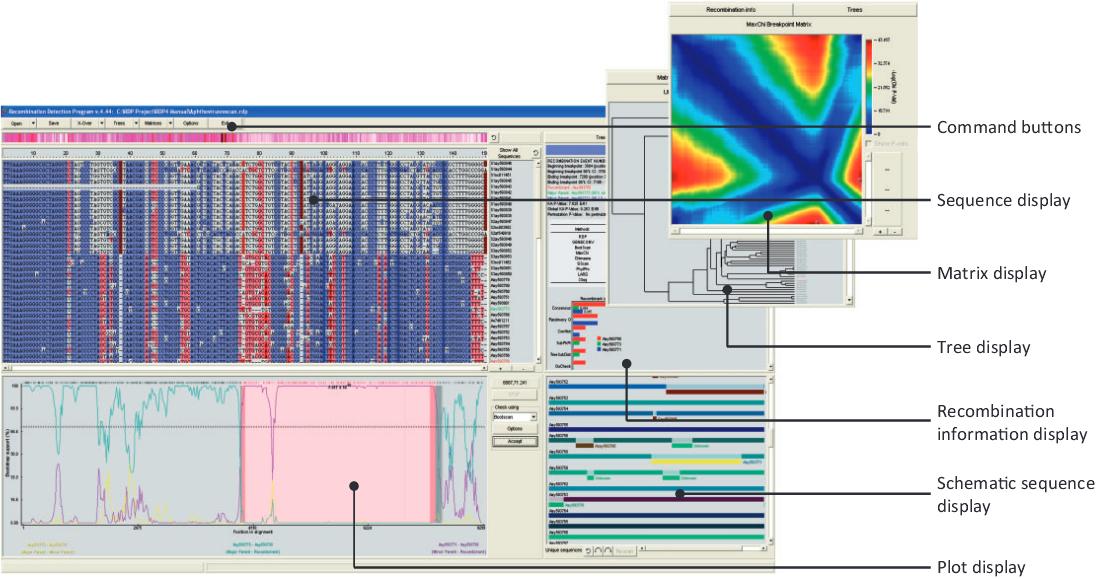
Recombination Detection Program) RDP) نرم افزاری برای شناسایی و آنالیز نوترکیبی در همردیف های DNA است. از آنجاییکه نوترکیبی ژنتیکی یک فرآیند تکاملی مهم در بسیاری از انواع مختلف ویروس هاست، کاربرد این نرم افزار بیشتر در مطالعات ویروس شناسی است، اگرچه در ژنوم های دیگر موجودات مانند انسان و باکتری هم کاربرد دارد. روش های مورد استفاده شامل RDP، MaxChi، Chimaera، Geneconv، Bootscan، Siscan، 3SEQ، VisRD و BURT می باشند. فرمت های مختلف فایل همردیفی از جمله PHYLIP، GDE، FASTA، CLUSTAL، GCG، NEXUS، MEGA و DNAMAN برای این نرم افزار قابل شناسایی و باز کردن هستند.
از قابلیت های این نرم افزار می توان به موارد زیر اشاره کرد:
قرائت مکان های ORF و نام ها ازفایل های بانک اطلاعاتی GenBank و یا از فایل های متنی
نمایش گرافیکی توالی
نمایش اطلاعات نوترکیبی
نمایش پلات نوترکیبی (شامل نمایش مکان های متغیر نوکلئوتیدی، مکان های شکست نوترکیبی و ...)
نمایش درخت فیلوژنتیکی بر اساس الگوریتم های UPGMA، NJ، FatNJ، ML، Bayesian
ترسیم انواع مختلف ماتریس
و روش های تکمیلی دیگر
این نرم افزار را می توانید بصورت رایگان از لینک زیر دانلود نمایید:
http://web.cbio.uct.ac.za/~darren/RDP4.95BetaBig.exe

Gene Runner نرم افزاری بسیار خوب و رایگان برای آنالیز توالی DNA است. از قابلیت های مهم این نرم افزار می توان به موارد زیر اشاره کرد:
فراهم کردن اطلاعات مفید و خصوصیات توالی (annotation)
جستجوی توالی های DNA برای مکان های برشی
جستجوی توالی های پروتئینی برای مکان های برشی پپتیداز
ترجمه DNA به توالی پروتئین و جستجوی ORF
طراحی پرایمر برای PCR و توالی یابی
امکان ترازبندی چندگانه توالی (multiple sequence alignments)
این نرم افزار را می توانید بصورت رایگان از لینک زیر دانلود نمایید:
http://www.generunner.net/GeneRunner.msi (برای ویندوز 32 بیتی)
http://www.generunner.net/GeneRunnerx64.msi (برای ویندوز 64 بیتی)
BioToolKit 320 این پکیج شامل مجموعه زیادی از نرم افزارهای مفید در حوزه زیست شناسی مولکولی است. ابزارهای متعددی برای PCR، طراحی پرایمر، پروتئین، برش های آنزیمی، ژنتیک کلاسیک، ژنومیکس، آزمون های آماری و .....
لیست همه نرم افزارهای موجود در این پکیج در لینک زیر قابل دسترسی هستند:
http://www.changbioscience.com/biotoolkit2.html
توجه: این مجموعه نرم افزاری، برای اجرا شدن نیاز به برنامه جاوا ( نسخه JRE 1.4 به بالا) دارد.
نسخه آزمایشی (Demo) را می توانید از لینک زیر دانلود نمایید.
http://www.changbioscience.com/btk320/
برای نسخه کامل این مجموعه نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)
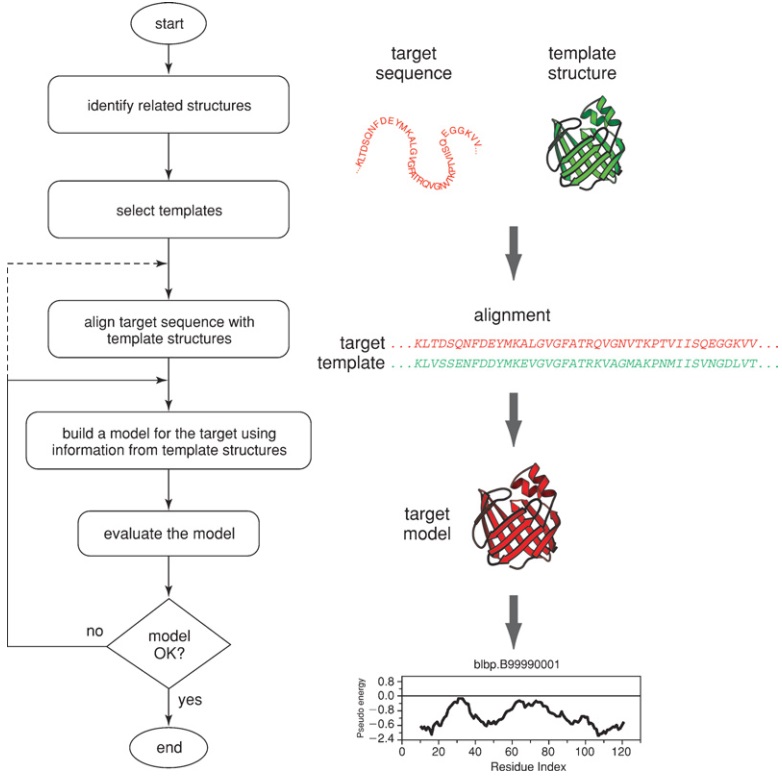
Modeller نرم افزاری برای مدلسازی مقایسه ای یا همولوژی ساختارهای سه بعدی پروتئین است. بدین منظور کاربر یک ردیف توالی را به نرم افزار می دهد تا مدلسازی با استفاده از ساختارهای شناخته شده مرتبط به آن توالی انجام شود و نرم افزار به طور خودکار یک مدل که شامل تمام اتم ها غیر از هیدروژن است را محاسبه می کند. این نرم افزار همچنین می تواند وظایف دیگری را نیز انجام دهد از جمله: مدل سازی لوپ ها در ساختارهای پروتئینی، بهینه سازی مدل های مختلف از ساختار پروتئین با توجه به تابع هدف تعریف شده، ترازبندی چندگانه توالی ها و یا ساختار های پروتئینی، خوشه بندی، جستجو توالی در پایگاه داده ها، مقایسه ساختارهای پروتئین و غیره .. استفاده کرد. نرم افزار روی سیستم عامل های ویندوز، Mac و لینوکس قابل نصب می باشد.
توجه: نرم افزار مبتنی بر نوشتن خط دستور (command line) است و گرافیکی نیست. از اسکریپت زبان برنامه نویسی پایتون (Python) برای اجرا استفاده می کند.
نسخه های گرافیکی آن بصورت تجاری در دسترس هستند که می توانید آن ها را از طریق لینک زیر بیابید:
https://salilab.org/modeller/wiki/Links
این نرم افزار را می توانید بصورت رایگان از لینک زیر دانلود نمایید:
https://salilab.org/modeller/download_installation.html
توجه: با آنکه نرم افزار رایگان می باشد، ولی بایستی در سایت آن فرم ثبت نام برای دریافت لایسنس را پرکنید تا بتوانید نرم افزار را نصب نمایید.
https://salilab.org/modeller/registration.html
راهنمای استفاده از نرم افزار با مثال های مختلف از طریق لینک زیر قابل دسترسی است:
https://salilab.org/modeller/tutorial/
https://salilab.org/modeller/9.19/manual.pdf
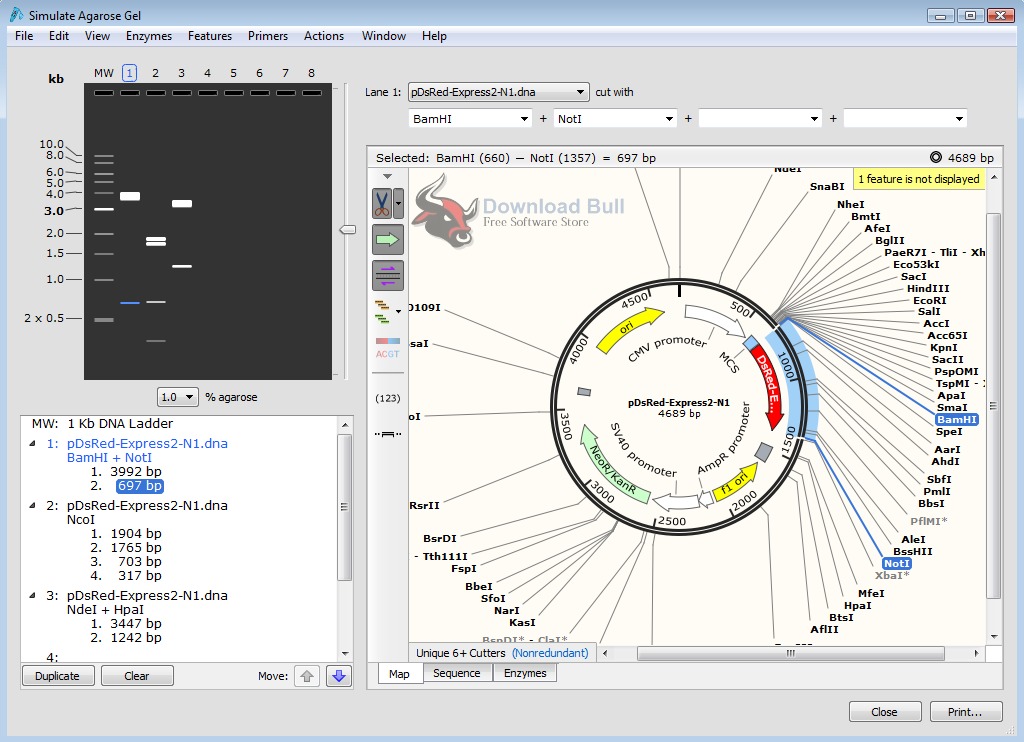
SnapGene یکی از نرم افزارهای بسیار خوب برای کار با توالی ها و سازه های DNA است. به کمک این نرم افزار می توان برنامه ریزی، شبیه سازی و مستندسازی فرآیندهای PCR و کلون سازی DNA را انجام داد. از ویژگی های این نرم افزار می توان به موارد زیر اشاره کرد:
شناسایی و مشاهده ORFها و مکان های اتصال پرایمر
طراحی پرایمر برای PCR، توالی یابی یا جهش زایی (Mutagenesis)
شبیه سازی کلون سازی برشی (restriction cloning) و دیگر انواع کلون سازی
شبیه سازی جهش زایی به کمک پرایمر
شبیه سازی الکتروفورز ژل آگارز
In-Fusion® Cloning
Gibson® Assembly
Gateway Cloning
TA or GC cloning
دسترسی به فایل های نقشه انواع پلاسمیدها بصورت آنلاین
اشتراک گذاری توالی های تفسیرشده با دیگر محققان
و ...
نسخه آزمایشی (30 روزه) این نرم افزار را میتوانید از لینک زیر دانلود نمایید:
http://www.snapgene.com/products/snapgene/free_trial/
برای نسخه کامل این نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)
sajjad.mansoury@yahoo.com
RNA ،miRNAsهایی با طول 24-19 نوکلئوتیدی هستند که ژن های هدفشان را بوسیله اتصال به mRNA های هدف تنظیم یا خاموش می کنند. miRNAها نقش های مهمی در فعالیت های مهم سلولی مانند رشد، مورفوژنز، آپوپتوزیز و تمایز ایفا می کنند.
RNAhybrid برنامه ای برای پیش بینی سریع و کارآمد هیبریداسیون توالی هدف با microRNA بر اساس حداقل انرژی آزاد (minimum free energy) است. این نرم افزار در سه نسخه آنلاین، از راه دور (از طریق WebService) و قابل دانلود (مبتنی بر دستور نویسی) ارائه شده است. در سایت نرم افزار ذکر شده است که نسخه قابل دانلود فاقد بعضی ویژگی ها است که در نسخه آنلاین موجود است. بنابراین پیشنهاد می شود از نسخه آنلاین سایت استفاده نمایید.
نسخه آنلاین آن از طریق لینک زیر قابل دسترس است:
https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid?id=rnahybrid_view_submission
نسخه قابل دانلود آن برای ویندوز را می توانید از لینک زیر دانلود نمایید:
http://bibiserv.cebitec.uni-bielefeld.de/resources/download/rnahybrid/RNAhybrid-Win7-2.1.2.tar.gz
راهنمای آموزشی اجرا و استفاده از نرم افزار به زبان فارسی:
https://www.dropbox.com/s/lfk8dkmqqsgeglq/RNAhybrid%20Persian.pdf?dl=0
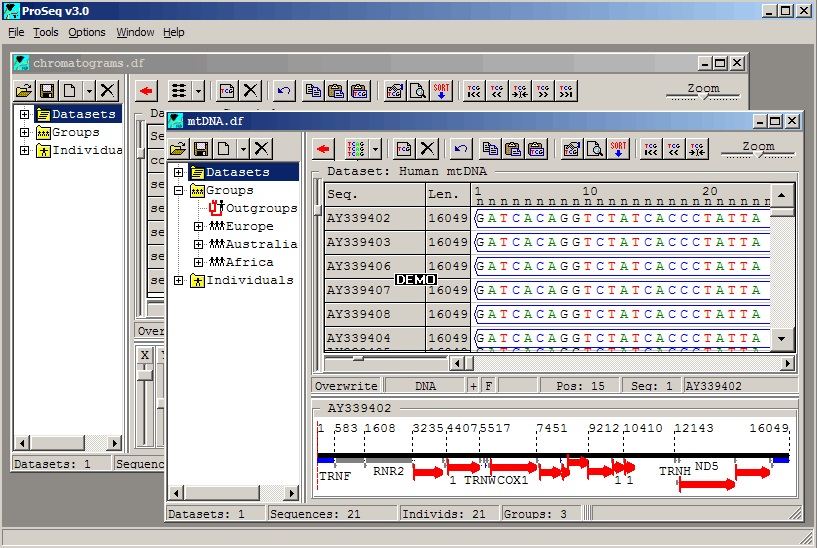
ProSeq نرم افزاری برای مدیریت و آنالیز ژنتیک جمعیت داده های پلی مورفیسم DNA است. این برنامه شامل یک ویرایشگر توالی/همردیفی و یک پایگاه اطلاعاتی داخلی است که آماده سازی و دست ورزی داده های پلی مورفیسم چندژنی را تسهیل می کند. نسخه جدید این برنامه می تواند داده های حجیم حاصل از توالی یابی را نیز پردازش کند.
این نرم افزار رایگان و کم حجم را می توانید از لینک زیر بصورت رایگان دریافت نمایید.
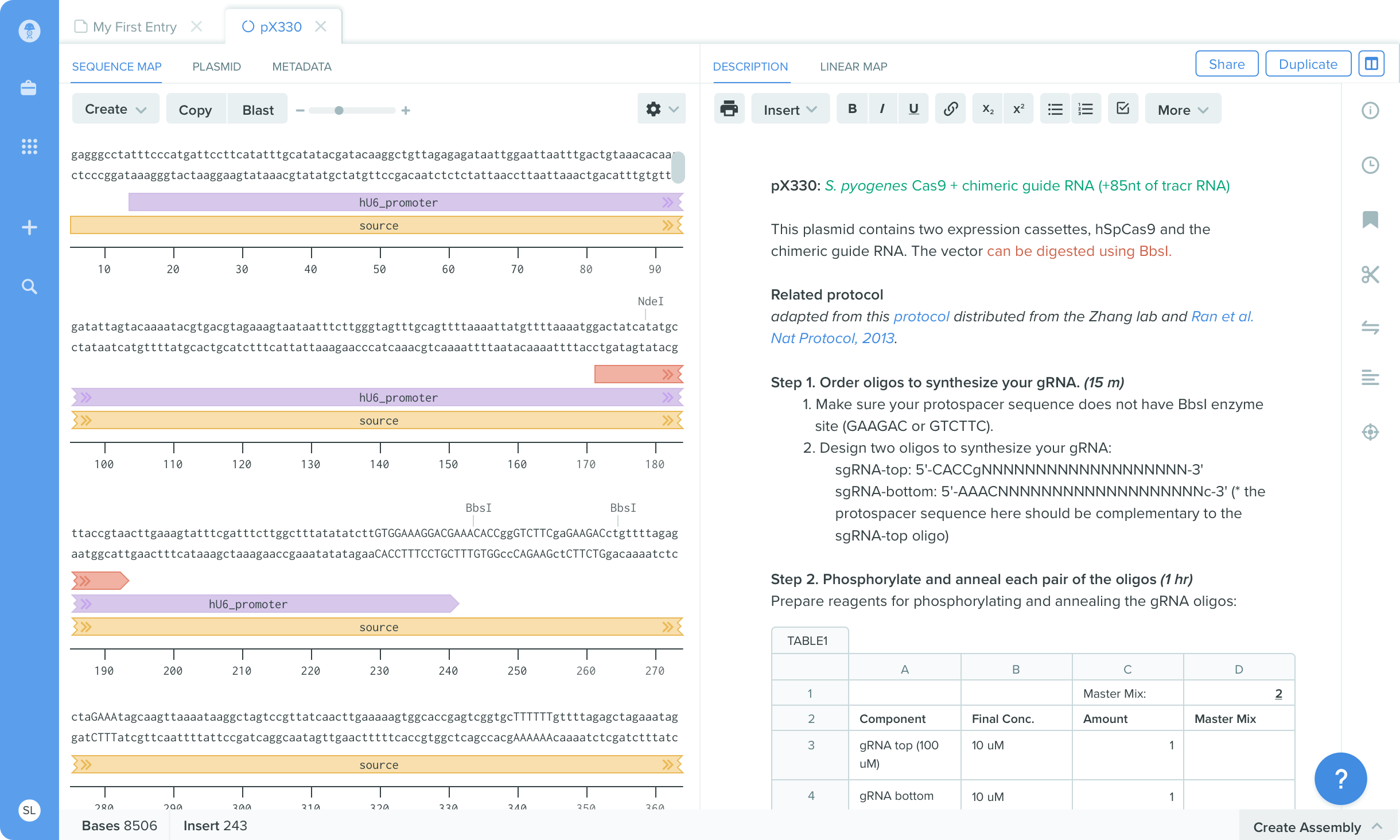
Benchling یک پلتفرم تحقیقاتی آنلاین است که محققان میتوانند آزمایشات مولکولی خود را طراحی، ثبت و به اشتراک بگذارند. ویژگی مهم این پلتفرم، آنالیز توالی ها بصورت اتوماتیک با بهترین ابزارهای این کار می باشد. از جمله آن ها می توان به تفسیر توالی ها (Annotation)، آنالیز آن ها با استفاده از آنزیم های برشی، کلون سازی، طراحی پرایمر، همردیف سازی توالی ها، طراحی و آنالیز RNA های راهنما (Guide RNAs) برای کاربرد در فناوری CRISPR، اسمبل کردن آن ها به داخل پلاسمیدها، طراحی الگوی نوترکیبی همولوگ و ... اشاره کرد.
توجه: برای استفاده از این پلتفرم بایستی ابتدا در سایت ثبت نام کرده و حساب کاربری ایجاد کنیم.
ورود به این پلتفرم با استفاده از لینک زیر:
راهنمای استفاده از ابزارهای مختلف پلتفرم هم در لینک زیر قرار دارد:
https://benchling.com/tutorials/
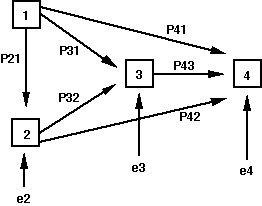
تجزیه علیت یا تجزیه مسیر (Path Analysis) آنالیزی است که برای تفکیک ضریب همبستگی (r) به اثرات مستقیم و غیرمستقیم متغیرهای مستقل (X) بر روی متغیر وابسته (Y) کاربرد دارد. PATH2 یک نرم افزار کم حجم و تحت DOS است که می تواند به راحتی این آنالیز را برای ما انجام دهد.
دانلود نرم افزار به همراه راهنمای فارسی
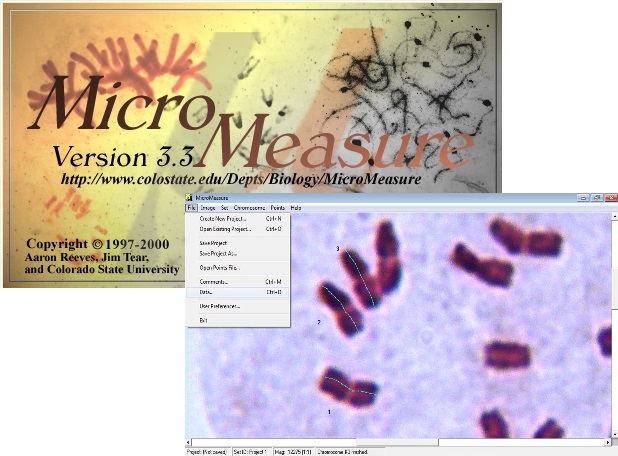
MicroMeasure نرم افزار سیتوژنتیکی آنالیز تصاویر میکروسکوپی کروموزوم هاست. این برنامه می تواند خصوصیات کروموزوم ها از جمله طول بازوهای بزرگ و کوچک، نسبت بازوها، نواحی یوکروماتین و هتروکروماتین و .. را بر اساس تعریف نقاط و نوع قطعه توسط کاربر و همچنین میزان بزرگ نمایی، ارائه کند. پس از محاسبه، خروجی داده ها بصورت اتوماتیک در یک فایل EXCEL ذخیره می شود.
نرم افزار قابلیت نصب رو سیستم عامل های ویندوز XP و 7 را دارد.
این نرم افزار رایگان و کم حجم را می توانید از لینک زیر بصورت رایگان دریافت نمایید.
همچنین دو نمونه فایل برای آشنایی و یادگیری کار با نرم افزار در سایت قرار داده شده است که می تواند از طریق لینک زیر به آن ها دسترسی داشته باشید:
http://sites.biology.colostate.edu/MicroMeasure/Help/samples.html

DNASTAR Lasergene Suite یکی از جامع ترین و کاربردی ترین پکیج های نرم افزاری در حوزه بیوانفورماتیک است که کاربردهای زیادی در تحقیقات ژنومیکس، زیست شناسی ساختاری و مولکولی دارد. در این نسخه جدید قابلیت های متعددی نسبت به نسخه های قبلی آن افزوده شده است. در زیر به برخی از آن ها اشاره می شود:
آنالیز توالی DNA
اسمبلی و آنالیز توالی های Next-gen
پروتئومیکس
و ...
برای نسخه کامل این نرم افزار با ایمیل زیر مکاتبه فرمایید (قیمت 50 هزار تومان)

مستند اول، مستند کوتاهی (حدود 30 دقیقه) راجع به بیوگرافی گریگور مندل، پدر علم ژنتیک است که می توانید از لینک زیر دریافت نمایید.
دانلود زیر نویس انگلیسی
مستند دوم، مستندی درباره بزرگترین کشفیات علم ژنتیک است که می توانید از لینک زیر دانلود کنید.
دانلود زیرنویس انگلیسی

Flapjack نرم افزاری برای مشاهده گرافیکی داده های ژنوتیپی و هاپلوتیپی است که می تواند داده هایی با حجم بالا (مانند داده های NGS) را نیز مدیریت کند. استفاده از این ابزار آنالیز این نوع داده ها را تسهیل می سازد. این نرم افزار امکان مقایسه بین مارکرها، لاین ها و کروموزوم ها را به صورت گرافیکی فراهم می کند. ورودی این نرم افزار داده های نقشه، ژنوتیپی و صفات ارزیابی شده است. از جمله ویژگی های مهم این نرم افزار می توان به موارد زیر اشاره کرد:
نمایش گرافیکی ژنوتیپ ها و آلل های آن ها با رنگ های مختلف با توجه به حالت، فراوانی یا تشابه
نمایش حذفیات (deletions) و درجیات (insertions) داده ها
مرتب کردن و گروه بندی لاین ها با توجه به تشابه ژنوتیپی آن ها با دیگر لاین ها
همترازی داده های نقشه (مانند مکان های QTL) با ژنوتیپ ها برای شناسایی هاپلوتیپ های مرتبط
و ...
این نرم افزار را میتوانید بصورت رایگان از لینک زیر دانلود نمایید:

STRUCTURE نرم افزاری برای استنتاج ساختار جمعیت (inferring population structure) بر اساس یک روش گروه بندی (Bayesian) مبتنی بر مدل (Ancestry Models) با استفاده از داده های ژنوتیپی مارکرهاست. این روش در مقاله پریتچارد و همکاران (2000) معرفی شد. از کاربردهای این نرم افزار می توان به اثبات حضور ساختار جمعیت، شناسایی جوامع ژنتیکی متمایز، انتساب افراد به جمعیت ها و شناسایی مهاجران (migrants) و افراد مخلوط شده (admixed individuals) اشاره کرد.
این نرم افزار می تواند روی سیستم عامل های ویندوز، لینوکس و مکینتاش اجرا شود.
این نرم افزار را می توانید بصورت رایگان از لینک زیر دریافت نمایید: