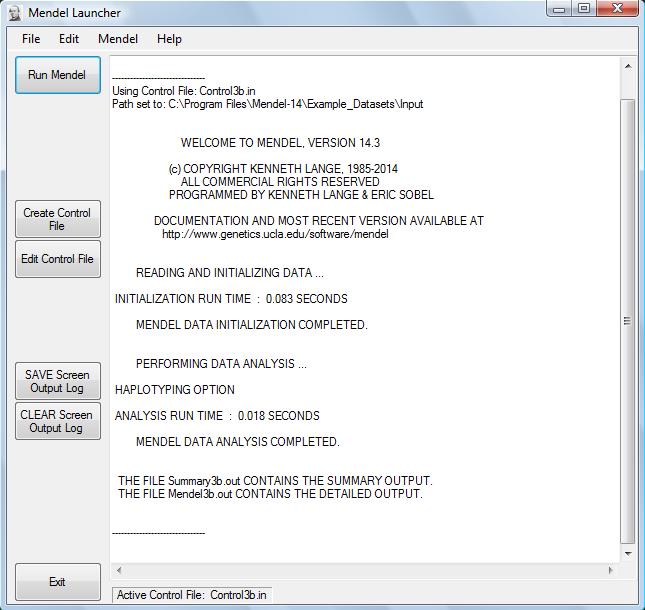
Mendel یک پکیج نرم افزاری جامع برای آنالیز آماری صفات کیفی و کمی از لحاظ ژنتیکی است. آپشن های این نرم افزار شامل موارد زیر می شود:
order and map a set of markers along a chromosome
use parametric linkage methods to localize a gene on a fixed marker map
use non-parametric linkage (NPL) methods to localize a gene on a fixed marker map
estimate pedigree haplotypes
identify potential genotyping errors
test for allelic association by the TDT or the gamete-competition model
estimate kinship coefficients conditional on marker data
perform genetic risk calculations
test for paternity or other pedigree relationships
estimate allele frequencies using either pedigrees or a random sample
estimate trait genotype penetrances
estimate ethnic composition using pedigree data
identify deviations from Hardy-Weinberg and linkage equilibrium
simulate genetic data by gene dropping
localize QTLs, including X-linked QTLs, using variance component analysis
perform association analysis on quantitative traits
test for association given linkage and
trim pedigrees to a core group and all necessary connecting relatives
dense SNP genotype imputation, including haplotypes
GWAS: dense SNP association testing, including rare variants
maternal-fetal genotype incompatibility test
inbred strain QTL analysis
simulate trait values incorporating major loci and various models
pedigree-based GWAS (SNP association testing)
این نرم افزار مثال های زیادی را در قالب فایل های ورودی و خروجی در خود دارد که می توان برای یادگیری از آن ها استفاده کرد.
راهنمای نرم افزار (به زبان انگلیسی)
دانلود راهنما
برای دانلود نرم افزار ابتدا بایستی در سایت http://www.genetics.ucla.edu/software/registration ثبت نام کنید و سپس با وارد کردن ایمیل خود در سایت نرم افزار را دانلود نمایید.















 مدیر وبلاگ:
مدیر وبلاگ: